El análisis del viroma total arroja luz sobre la materia oscura viral en las enfermedades inflamatorias crónicas del intestino
Artículo Comentado - Adulto
Por el Pr. Harry Sokol
Gastroenterología y nutrición, Hospital Saint-Antoine, París, Francia
Espacio para el público general
Encuentra aquí tu espacio dedicado
en_sources_title
en_sources_text_start en_sources_text_end
Capítulos
Bacteriophages infecting bacteria, illustration. A bacteriophage, or phage, is a virus that infects bacteria.
Acerca de este artículo
Autor
Comentario del artículo original de Clooney et al. (Cell Host & Microbe 2019) [1]
En la actualidad, se cree que el viroma intestinal humano tiene un impacto significativo en el microbioma y la salud humana. Sin embargo, la mayoría de los análisis se han llevado a cabo en una fracción limitada de virus conocidos. Al realizar un análisis del viroma completo sobre una cohorte publicada de enfermedad inflamatoria crónica intestinal (EICI) y de nuevos datos sobre la rectocolitis hemorrágica, los autores han puesto de manifiesto el componente viral de la microbiota intestinal humana en las EICI más allá de la minoría identificable. Los autores han observado cambios específicos en las EICI y un aumento del número de secuencias de bacteriófagos moderados en pacientes que padecen la enfermedad de Crohn. Contrariamente a los métodos anteriores que dependen de bases de datos, no se ha observado ningún cambio en la riqueza viral. En los pacientes que sufren una EICI, los cambios en la composición del viroma reflejaban alteraciones en la composición bacteriana. Además, la combinación de los datos del bacterioma y del viroma mejoraba la potencia de la clasificación entre salud y enfermedad. El planteamiento consistente en analizar genomas de virus enteros a través de varias cohortes revela señales significativas durante las EICI que podrían ser cruciales para el desarrollo de biomarcadores y tratamientos en el futuro.
¿Qué se sabe ya sobre este tema?
Probablemente, el viroma sea una de las principales fuerzas que conforman el microbioma intestinal humano, pero quizá sea también su componente menos comprendido. Está dominado por bacteriófagos (fagos), que desempeñan una función vital en numerosas comunidades microbianas al estimular la diversidad, ayudar a renovar los nutrientes y facilitar la transferencia horizontal de genes. Comprender la función de los bacteriófagos en las estructuras de las comunidades microbianas será fundamental para comprender o controlar las alteraciones del microbioma intestinal humano asociadas a numerosas enfermedades Existen numerosas bacterias intestinales (y huéspedes potenciales de bacteriófagos) que son difíciles de cultivar, lo que significa que el análisis del viroma depende en gran medida de la secuenciación metagenómica y de los métodos bioinformáticos.
Puntos clave
-
No es posible estudiar una gran mayoría del viroma intestinal dado que no se encuentra en las bases de datos.
-
Un método independiente de las bases de datos desarrollado aquí permite comprender el viroma intestinal en su globalidad.
-
El viroma intestinal se encuentra alterado en las EICI, es menos estable y está dominado por bacteriófagos moderados.
-
El viroma intestinal podría utilizarse como biomarcador u objetivo terapéutico en el futuro.
Sin embargo, no existen genes marcadores universales para los bacteriófagos (como el ARNr 16S para las bacterias) y hay una carencia de información taxonómica, por culpa de bases de datos pobres, lo que significa que es necesario contar con métodos independientes de estas bases de datos. Los primeros estudios metagenómicos han puesto de relieve la diversidad del viroma intestinal humano, pero solo han sido capaces de clasificar una fracción menor (un 2 %) del ADN secuenciado [2]. Las mejoras en las tecnologías de secuenciación de alto rendimiento han permitido analizar el viroma con un inaudito nivel de detalle. Se ha confirmado que el viroma está increíblemente diversificado, que la mayoría no se alinea con las secuencias conocidas de las bases de datos (denominada la materia oscura viral), y que la composición es única en cada individuo.
Aunque el origen de las EICI sigue sin estar claro, estas enfermedades son multifactoriales y se asocian a alteraciones del microbioma intestinal. Ha aparecido un conjunto de datos que demuestra que el viroma intestinal se encuentra alterado en las EICI [3], con una mayor diversidad global y una mayor abundancia relativa del orden de los Caudovirales. Sin embargo, casi todos los resultados se han obtenido a partir de los cambios de composición de la fracción identificable del viroma, que representa tan solo el 15 % de los datos [3]. Esto limita la comprensión global del viroma y obstaculiza la identificación de los biomarcadores potenciales de la enfermedad. Es fundamental hallar un método de análisis independiente de la base de datos si se desea caracterizar plenamente los cambios del viroma intestinal.
¿Cuáles son los principales resultados aportados por este estudio?
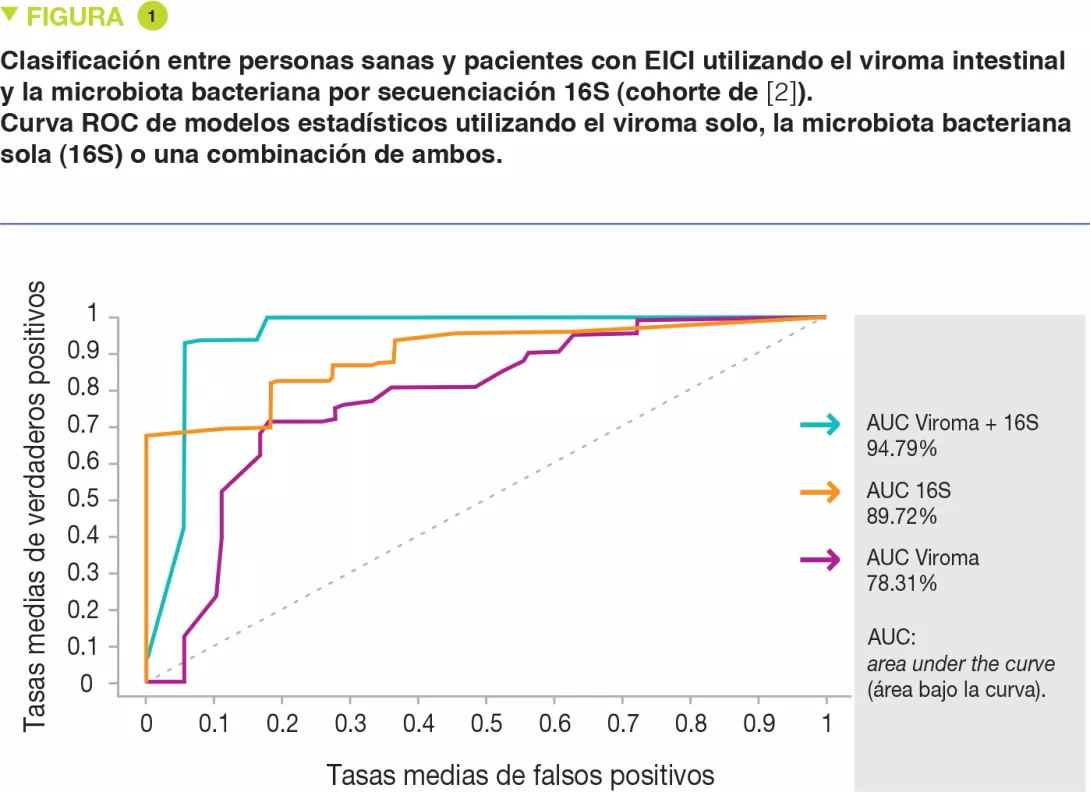
En el marco de este estudio, los autores han vuelto a analizar un conjunto de datos clave publicados anteriormente [3] y obtenidos a partir de pacientes con enfermedad de Crohn y rectocolitis hemorrágica y de pacientes control sanos. El problema del alto nivel de variación entre individuos se superó utilizando la homología de las proteínas y un algoritmo particular (clúster de Markov) para reagrupar las secuencias virales en rangos taxonómicos supuestos. De esta manera, ha sido posible describir los cambios de composición a través del viroma completo más allá de la minoría conocida. Los autores proponen que, contrariamente al viroma común de personas sanas constituido por bacteriófagos virulentos, el viroma de los pacientes con EICI está alterado, es menos estable y está dominado por bacteriófagos moderados. Han demostrado que las alteraciones del viroma imitan las del bacterioma y que cuando se utilizan conjuntamente, son más eficaces para diferenciar a los pacientes con EICI de las personas sanas (Figura 1). Los resultados se han validado en una cohorte longitudinal de pacientes con RCH. Este planteamiento, independiente de las bases de datos, podría utilizarse para esclarecer la materia oscura viral de numerosos estudios ya publicados.
¿Cuáles son las consecuencias en la práctica?
Estos resultados confirman que el viroma intestinal está alterado en las EICI y que puede asociarse a elementos de la microbiota bacteriana: podría utilizarse como biomarcador diagnóstico e incluso pronóstico. Por otro lado, las alteraciones observadas sugieren que el viroma podría ser parcialmente responsable de las alteraciones de la microbiota bacteriana observada en las EICI. Por tanto, en el futuro, el viroma intestinal podría ser un objetivo o una herramienta terapéutica para las EICI.
Conclusión
El viroma intestinal se estudia en su globalidad utilizando un método independiente de las bases de datos, que solamente permiten analizar una pequeña parte. Mientras que el viroma común de personas sanas está constituido por bacteriófagos virulentos, el viroma de pacientes con EICI está alterado, es menos estable y está dominado por bacteriófagos moderados. El viroma asociado a la microbiota bacteriana permite diferenciar a los pacientes con EICI de las personas sanas con más eficacia que utilizando exclusivamente el viroma o la microbiota bacteriana. Estos resultados abren nuevas perspectivas para el uso del viroma como biomarcador u objetivo terapéutico en las EICI.