L’analyse du virome total éclaire la matière noire virale dans les maladies inflammatoires chroniques de l’intestin
Article commenté - Rubrique adulte
Pr. Harry Sokol
Gastro-entérologie et nutrition, Hôpital Saint-Antoine, Paris, France
Section grand public
Retrouvez ici votre espace dédié
en_sources_title
en_sources_text_start en_sources_text_end
Chapitres
Bacteriophages infecting bacteria, illustration. A bacteriophage, or phage, is a virus that infects bacteria.
A propos de cet article
Auteur
Commentaire de l’article original de Clooney et al. (Cell Host & Microbe 2019) [1]
On pense actuellement que le virome intestinal humain a un impact significatif sur le microbiote et la santé humaine. Cependant, la plupart des analyses ont été effectuées sur une fraction limitée de virus connus. En réalisant une analyse du virome entier sur une cohorte publiée de maladie inflammatoire chronique de l’intestin (MICI) et des nouvelles données sur la rectocolite hémorragique, les auteurs ont mis en lumière la composante virale du microbiote intestinal humain dans les MICI au-delà de la minorité identifiable. Les auteurs y ont observé des changements spécifiques au cours des MICI et une augmentation du nombre de séquences de phages tempérés chez les patients atteints de la maladie de Crohn. Contrairement aux méthodes antérieures dépendantes de la base de données, aucun changement dans la richesse virale n’a été observé. Chez les sujets atteints de MICI, les changements dans la composition du virome reflétaient des altérations de la composition bactérienne. De plus, l’association des données du bactériome et du virome améliorait la puissance de classification entre la santé et la maladie. Cette approche de l’analyse des génomes de virus entiers à travers plusieurs cohortes met en évidence des signaux significatifs au cours des MICI, qui pourraient être cruciaux pour le développement de futurs biomarqueurs et traitements.
Que sait-on déjà à ce sujet ?
Le virome est probablement l’une des principales forces qui façonnent le microbiote intestinal humain, mais il s’agit peut-être aussi de sa composante la moins bien comprise. Il est dominé par les bactériophages (phages), qui jouent un rôle vital dans de nombreuses communautés microbiennes en stimulant la diversité, en aidant au renouvellement des nutriments et en facilitant le transfert horizontal de gènes. Comprendre le rôle des bactériophages dans les structures des communautés microbiennes sera essentiel si nous voulons comprendre ou contrôler les altérations du microbiome intestinal humain qui sont associées à de nombreuses maladies. De nombreuses bactéries intestinales (et des hôtes potentiels de phages) restent difficiles à cultiver, ce qui signifie que l’analyse du virome dépend fortement du séquençage métagénomique et des approches bio-informatiques.
Points clés
-
Une large majorité du virome intestinal ne peut être étudié car il est absent des bases de données.
-
Une méthode indépendante des bases de données développée ici permet d’appréhender le virome intestinal dans sa globalité.
-
Le virome intestinal est altéré dans les MICI, moins stable et dominé par des phages tempérés.
-
Le virome intestinal pourrait être utilisé comme biomarqueur ou cible thérapeutique dans le futur.
Cependant, il n’existe pas de gènes marqueurs universels pour les phages (similaires à l’ARNr 16S pour les bactéries) et il y a un manque d’informations taxonomiques avec des bases de données pauvres, ce qui signifie que des méthodes indépendantes des bases de données sont nécessaires. Les premières études métagénomiques ont mis en évidence la diversité du virome intestinal humain, mais n’ont pu classer qu’une fraction mineure (2 %) de l’ADN séquencé [2]. Les améliorations des technologies de séquençage à haut débit ont permis au virome d’être analysé avec un niveau de détail inédit. Il a été confirmé que le virome est incroyablement diversifié, que la majorité ne s’aligne pas sur les séquences connues dans les bases de données (appelée la matière noire virale), et que la composition est unique aux individus..
Bien que l’étiologie des MICI reste floue, ces maladies sont multifactorielles et associées à des altérations du microbiome intestinal. Un ensemble de données émergent prouvant que le virome intestinal est altéré dans les MICI [3] avec une diversité globale accrue et une abondance relative accrue de l’ordre des Caudovirales. Pourtant, presque tous les résultats ont été tirés des changements de composition de la fraction identifiable du virome, qui peuvent représenter aussi peu que 15 % des données [3]. Cela limite la compréhension globale du virome et entrave l’identification des biomarqueurs potentiels de la maladie. Une méthode d’analyse indépendante de la base de données est essentielle si l’on souhaite caractériser pleinement les changements du virome intestinal.
Quels sont les principaux résultats apportés par cette étude ?
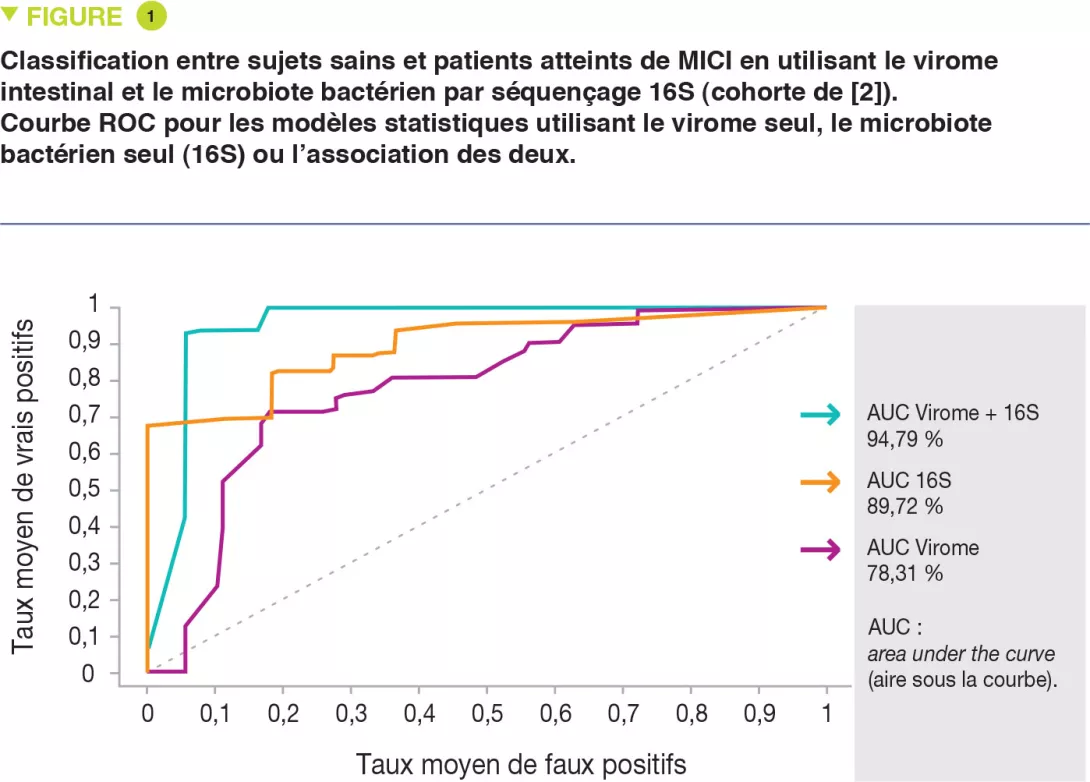
Dans le cadre de cette étude, les auteurs ont réanalysé un ensemble de données- clés publiées précédemment [3] obtenues à partir de sujets avec maladie de Crohn et rectocolite hémorragique et des témoins sains. Le problème du niveau élevé de variation interindividuelle a été surmonté en utilisant l’homologie des protéines et un algorithme particulier (cluster de Markov) pour regrouper les séquences virales en rangs taxonomiques supposés. De cette façon, il a été possible de décrire les changements de composition à travers le virome entier au-delà de la minorité connue. Les auteurs proposent que, contrairement au core virome des sujets sains constitué de phages virulents, le virome des patients atteints de MICI est altéré, moins stable et dominé par des phages tempérés. Ils montrent que les altérations du virome miment celles du bactériome et que lorsqu’elles sont utilisées ensemble, elles offrent une meilleure efficacité pour différencier les patients atteints de MICI de sujets sains (Figure 1). Les résultats ont été validés dans une cohorte longitudinale de patients atteints de RCH. Cette approche, indépendante des bases de données, pourrait être utilisée pour éclairer la matière noire virale de nombreuses études déjà publiées.
Quelles sont les conséquences en pratique ?
Ces résultats confirment que le virome intestinal est altéré dans les MICI et que celui- ci peut être associé à des éléments du microbiote bactérien ; il pourrait être utilisé comme biomarqueur diagnostique, voire pronostique. D’autre part, les altérations observées suggèrent que le virome pourrait être en partie responsable des altérations du microbiote bactérien observé dans les MICI. Dans ce sens, le virome intestinal pourrait donc être une future cible ou un futur outil thérapeutique dans les MICI.
Conclusion
En utilisant une méthode indépendante des bases de données, le virome intestinal est étudié dans sa globalité alors qu’habituellement, seule une minorité est analysée. Alors que le core virome des sujets sains est constitué de phages virulents, le virome des patients atteints de MICI est altéré, moins stable et dominé par des phages tempérés. Le virome associé au microbiote bactérien permet de différencier les patients atteints de MICI de sujets sains de manière plus efficace qu’en utilisant exclusivement le virome ou le microbiote bactérien. Ces résultats ouvrent de nouvelles perspectives pour l’utilisation du virome comme biomarqueur ou cible thérapeutique dans les MICI.