Microbioma intestinal e resistência antimicrobiana
Pelo Dr. Joseph Nesme
Professor Assistente, Microbiologia, Departamento de Biologia,Universidade de Copenhaga, Dinamarca
Área para o público geral
Encontre aqui o seu espaço dedicado
en_sources_title
en_sources_text_start en_sources_text_end
Capítulos
Sobre este artigo
A resistência aos antibióticos é antiga e prevalecente e o resistoma ambiental é um reservatório de genes de resistência que alimenta a pandemia silenciosa de resistência antimicrobiana (RA). A transferência horizontal de genes, a dinâmica populacional e a co-seleção aceleram o aparecimento e a disseminação do resistoma e, em 2050, mais de 10 milhões de mortes por ano dever-se-ão à resistência aos antibióticos. A colonização precoce do microbioma intestinal é propensa a disbioses que podem levar a um aumento da abundância e diversidade do resistoma intestinal dos bebés. A adoção de uma abordagem “Uma Só Saúde”, que considere a interligação entre a saúde humana, a saúde animal e o ambiente, é essencial para enfrentar os desafios complexos colocados pela RA.
Resistência aos antibióticos no ambiente: Um desafio pré-existente
A resistência aos antibióticos (RA) é um fenómeno antigo e prevalecente no ambiente, existente muito antes da introdução de moléculas de antibióticos como terapêutica. O ambiente funciona como um vasto reservatório de genes de resistência aos antibióticos, com diversas comunidades microbianas que albergam mecanismos de resistência. A RA foi encontrada em vários contextos ambientais, incluindo o solo, a água, as plantas, os animais e até mesmo no permafrost ártico com 30 000 anos [1, 2]. O papel ecológico das moléculas antibióticas e da resistência associada em contextos não clínicos permanece pouco claro, mas realça o facto de um conjunto de genes prontamente disponíveis ser anterior à utilização clínica de antibióticos e explicar a rápida emergência de agentes patogénicos. A atual crise dos antibióticos é um fenómeno evolutivo e as estratégias de atenuação devem ter em conta a ecologia microbiana. O que é problemático é a rápida aquisição de resistência por parte de agentes patogénicos anteriormente sensíveis, que conduz a falhas terapêuticas, especialmente quando se espera que muito poucos novos antimicrobianos cheguem ao mercado [3].
Mecanismos que determinam o aparecimento e a disseminação do resistoma
O resistoma refere-se ao conjunto completo de genes que codificam as proteínas relacionadas com a resistência aos antibióticos (RA) ou as proteínas que podem potencialmente evoluir para poderosos agentes de RA [4] (figura 1). Isto inclui genes de RA reconhecidos em bactérias patogénicas (as problemáticas), genes de RA de organismos produtores de antibióticos, como Streptomyces spp. que produzem o antibiótico estreptomicina e o gene de resistência associado [5], genes de RA crípticos (ou seja, genes que poderiam proporcionar resistência num contexto genético diferente; por exemplo, bombas de efluxo reguladas positivamente ou porinas reguladas negativamente) e genes de RA precursores que codificam proteínas com um nível mínimo de afinidade ou resistência a compostos antibióticos.
É de salientar que uma parte considerável destes subconjuntos de genes se sobrepõe devido à homologia das sequências, indicando que provavelmente têm um passado evolutivo partilhado.
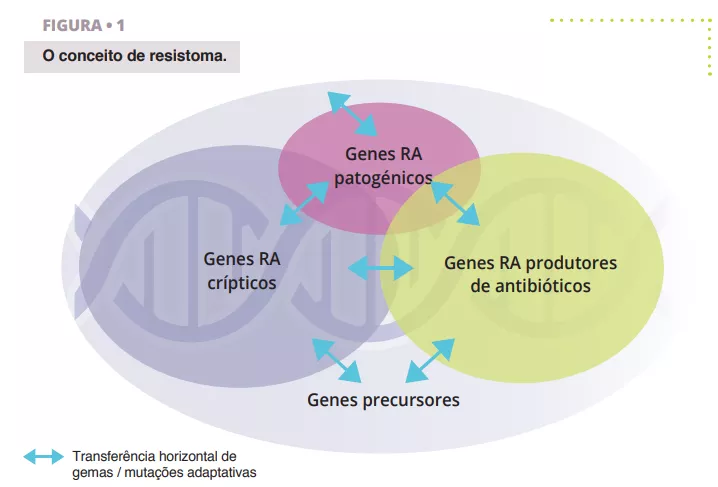
A noção de resistoma é distinta de “RA funcional e clinicamente relevante”. De facto, os genes do resistoma podem transitar entre os diferentes estados descritos acima por transferência horizontal de genes (HGT), mutações pontuais ou recombinação que conduzem a novos hospedeiros ou contextos genéticos onde o fenótipo de RA clinicamente relevante pode ser expresso. Por conseguinte, um gene de resistência não é problemático por si só, uma vez que depende do hospedeiro e do contexto genético, mas todos os genes do resistoma são uma ameaça potencial com diferentes resultados de risco para a saúde pública associados. A descoberta de um gene de resistência a uma molécula clinicamente relevante, localizado num elemento móvel e alojado num agente patogénico humano, constitui um risco crítico, mas o mesmo gene, ou homólogos próximos, encontrado numa bactéria não patogénica do solo e não associado a um elemento genético móvel, constitui um ARG (Gene de Resistência Antimicrobiana) de risco muito baixo. A classificação do risco de resistência aos antibióticos em estudos de resistoma é, por conseguinte, de importância primordial.
A transferência horizontal de genes (HGT) é um mecanismo fundamental responsável pela rápida disseminação de genes AR entre bactérias, mesmo entre linhagens distantes. Por exemplo, Bacteroides spp., um grupo predominante na microbiota intestinal humana, possui um gene de resistência aos macrólidos ermB idêntico ao encontrado em vários isolados de Clostridium perfringens, Streptococcus pneumoniae e Enterococcus faecalis de várias origens geográficas, indicando uma ligação genética entre Bacteroides e algumas bactérias Gram-positivas que não são predominantes no intestino humano [6]. Os elementos genéticos, como os plasmídeos, facilitam a transferência de genes de resistência entre diferentes espécies microbianas [7]. A HGT permite a disseminação de genes em diversos ambientes e populações bacterianas, contribuindo para a prevalência e diversidade globais da RA. A co-seleção é outro fator significativo na propagação da RA. A utilização de compostos não antibióticos, como os metais pesados e os biocidas, pode co-selecionar genes de RA, exercendo pressões seletivas sobre as populações microbianas, quer por co-resistência (diferentes determinantes de resistência presentes no mesmo elemento genético), quer por resistência cruzada (o mesmo determinante genético responsável pela resistência aos antibióticos e aos metais) [8]. A exposição a compostos antimicrobianos naturais, tais como os produzidos por microrganismos concorrentes ou quaisquer compostos co-seletivos, pode conduzir à seleção de estirpes resistentes [9]. A presença de antibióticos no ambiente, quer de fontes naturais quer de atividades humanas, contribui ainda mais para a pressão de seleção da resistência. Além disso, a utilização de antibióticos na agricultura e nas práticas veterinárias pode levar à contaminação do ambiente, promovendo o aparecimento e a disseminação de genes de resistência ambiental aos antibióticos ao longo do tempo [10].
A compreensão das barreiras e tendências à transferência de vetores genéticos que albergam genes de resistência é crucial para conceber estratégias de mitigação eco-evolutivas informadas para limitar a disseminação da RA no início da vida e, em geral, em contextos clínicos.
Disbiose e o resistoma intestinal nos bebés: Um equilíbrio delicado
A diversidade do reservatório ambiental de genes AR e o seu potencial de transferência representam uma ameaça para o microbioma intestinal humano no início da vida. Estratégias como a melhoria do tratamento de águas residuais, a utilização responsável de antibióticos na agricultura e na medicina veterinária e a redução da contaminação ambiental com resíduos de antibióticos e bactérias resistentes a antibióticos podem ajudar a atenuar a propagação da resistência [11]. Além disso, a monitorização e a vigilância dos reservatórios ambientais podem fornecer informações valiosas sobre a emergência e a persistência da RA e informar as intervenções de saúde pública.
A adoção de uma abordagem “Uma Só Saúde”, que considere a interligação entre a saúde humana, a saúde animal e o ambiente, é essencial para enfrentar os desafios complexos colocados pela RA.
O nosso intestino é rapidamente colonizado após o nascimento por microrganismos adquiridos das suas mães e do ambiente que as rodeia. É durante os primeiros anos de vida que as mudanças são drásticas e caracterizadas por uma baixa resiliência em comparação com o microbioma intestinal do adulto saudável, que é mais estável. Os recém-nascidos e os bebés são, portanto, mais propensos a perturbações nas comunidades microbianas, conhecidas como disbiose. Durante esse período, muitos fatores podem influenciar e perturbar a maturação intestinal e, potencialmente, ter consequências a longo prazo para a saúde [12]. Estudos em ratos mostraram que, durante esta janela crítica de desenvolvimento, mais do que um efeito direto das moléculas de antibióticos, é a alteração da composição da microbiota intestinal que desencadeia consequências metabólicas, como a obesidade [13].
Desvendar o resistoma dos antibióticos no intestino dos bebés: Informações de um grande estudo de coorte
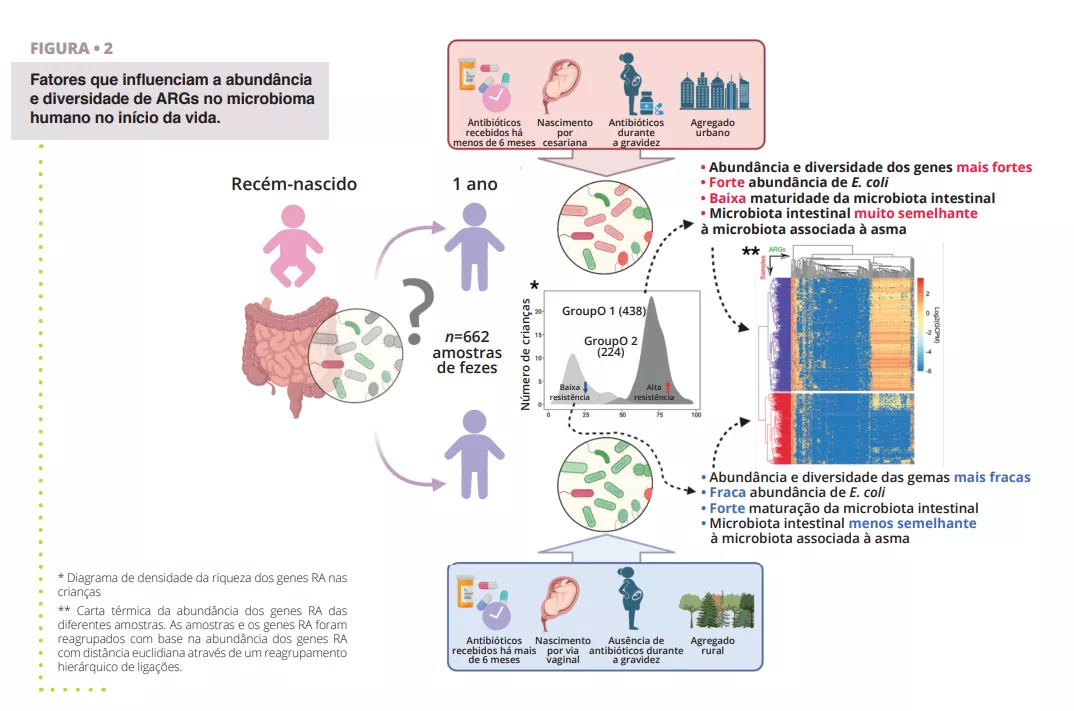
Embora a resistência aos antibióticos seja problemática em todas as idades da vida, o estabelecimento do microbioma intestinal em idades precoces representa uma janela de oportunidade para limitar a acumulação de um reservatório de genes AR no intestino. Por conseguinte, é importante identificar os vários fatores que aumentam ou reduzem a abundância de genes AR que podem propagar-se a agentes patogénicos infeciosos e causar falhas na terapia antibiótica ao longo da vida. Para estudar o resistoma global do intestino humano, os investigadores baseiam-se em abordagens holísticas que interrogam tanto a presença de espécies como o potencial funcional dos genomas, incluindo o resistoma aos antibióticos. Os investigadores baseiam-se na extração de ADN ambiental a partir de amostras de substituição (por exemplo, amostras de fezes para o intestino), seguida de sequenciação de alto rendimento não orientada (metagenómica shotgun). Cerca de 80% das espécies bacterianas do intestino humano detetadas por ferramentas moleculares não são cultiváveis, especialmente os anaeróbios especializados que habitam o intestino. É provável que muitos microrganismos estejam organizados em agregados celulares multi-espécies com co-dependências metabólicas que tornam o isolamento de estirpes puras delicado, se não impossível. No entanto, utilizando métodos computacionais, é possível reconstruir genomas quase completos a partir de metagenomas e associar genes codificados a espécies específicas ou mesmo a estirpes de bactérias. Num estudo recente, os investigadores analisaram amostras fecais de 662 bebés de uma coorte que acompanhou crianças desde o nascimento até aos 7 anos de idade [14]. Os objetivos do estudo eram estabelecer uma visão geral à escala da coorte do resistoma com 1 ano de idade e identificar fatores perinatais e ambientais associados à abundância e diversidade de ARG. Os investigadores utilizaram a sequenciação metagenómica shotgun de amostras obtidas com 1 ano de idade das 662 crianças para identificar ARGs e taxa bacterianos presentes nas amostras (figura 2).
Utilizando o seu grande conjunto de dados, os autores conseguiram reconstruir Genomas Associados a Metagenomas (MAGs), permitindo-lhes anotar com confiança a taxonomia dos genomas recuperados e o conteúdo dos seus genes AR. Um primeiro resultado observado foi que todas as crianças tinham pelo menos um tipo de ARG de múltiplos fármacos no seu intestino, indicando que, mesmo na ausência de tratamento com antibióticos, existe um resistoma residente associado ao microbioma intestinal. De facto, muitos genes de resistência a múltiplos fármacos foram identificados como bombas de efluxo. Estas proteínas são componentes normais de todas as células bacterianas, mas algumas podem conferir RA e são muito facilmente co-selecionadas por resistência cruzada, por exemplo, em relação a metais pesados ou biocidas, explicando potencialmente a sua elevada abundância no intestino, mas também em todos os ambientes [8, 15]. Outro resultado surpreendente foi a separação clara da coorte em dois grupos com base no seu perfil de resistoma. O primeiro grupo foi caracterizado por uma maior diversidade e abundância relativa de ARGs, com Escherichia coli como o principal contribuinte de ARGs, conforme mostrado na figura 2. Este facto está de acordo com observações anteriores de que as Enterobacteriaceae são abundantes no início da vida, mas devem diminuir rapidamente quando a população de Bacteroidetes começa a colonizar o intestino. A alteração desta maturação em algumas crianças pode estar associada a uma combinação de vários fatores, tais como a utilização de antibióticos, o modo de parto, o agregado familiar rural ou urbano, que parece atrasar a redução da população de Escherichia coli e levar a um aumento do resistoma com 1 ano de idade. Isto também é confirmado pela observação de que a maior abundância de ARGs está associada a uma menorpontuação de maturidade do microbioma intestinal, com base em rácios de taxa específicos relacionados com a idade [16].
O tratamento com antibióticos durante a gravidez e o primeiro ano de vida teve um impacto significativo na abundância do resistoma intestinal do bebé. Foi possível observar uma tendência temporal clara entre o tempo (mais de 6 meses ou menos) decorrido desde o último tratamento com antibióticos efetuado por uma criança e a abundância de ARGs no seu intestino.
Isto indica um certo nível de resiliência numa idade precoce que poderia ser potencialmente melhorado com uma intervenção direcionada, por exemplo, com pró- ou prebióticos, e que continua por testar. À escala da coorte, as maiores diferenças na abundância de ARG foram, no entanto, encontradas para genes de resistência a antibióticos que não foram prescritos às crianças, o que indica que os fatores perinatais e ambientais, para além da terapia antibiótica, também estão a determinar o resistoma intestinal. Outra observação desse estudo que liga o ambiente circundante, o resistoma associado e o resistoma intestinal foi o facto de as crianças cujos agregados familiares se situavam em zonas urbanas terem uma carga significativamente mais elevada de ARGs do que as crianças que viviam em zonas rurais. Há uma miríade de potenciais fatores de confusão que poderiam explicar este facto, mas reforça a ideia de que a contribuição do ambiente para o desenvolvimento do microbioma no início da vida é extremamente importante.
Pode colocar-se a hipótese de que a vida urbana está associada a um menor contacto com o exterior e a uma menor diversidade do microbioma, ou que o tipo de alojamento encontrado em ambiente rural (casa) ou urbano (apartamento) está associado a consequências no microbioma interior, tal como se observa no pó da cama.
Conclusão
A resistência aos antibióticos é antiga e prevalecente e o resistoma ambiental é um reservatório de potenciais genes de resistência clínica. Mecanismos como a transferência horizontal de genes, a intrincada dinâmica populacional e os efeitos de co-seleção foram identificados como aceleradores significativos da evolução e disseminação do resistoma aos antibióticos, tornando estirpes de agentes patogénicos anteriormente sensíveis agora resistentes à terapia antibiótica. O microbioma intestinal humano e a sua fase inicial de desenvolvimento são propensos à disbiose e à potencial invasão, seleção e co-seleção de bactérias portadoras de genes de resistência, o que pode ter consequências a longo prazo para a saúde. Não é claro até que ponto estas estão relacionadas com futuras terapias antibióticas falhadas. São necessários estudos multiómicos de longa duração e de alta resolução temporal para identificar, em primeiro lugar, padrões longitudinais de desenvolvimento do microbioma intestinal e, em segundo lugar, as vias mais prováveis de transferência de genes AR por HGT. Em conjunto, estes resultados fornecerão informações que permitirão conceber estratégias preventivas para minimizar o desenvolvimento de RA logo no início da vida e limitar a transferência para agentes patogénicos problemáticos. Este esforço é da maior importância para reduzir o peso da pandemia mundial de RAM e melhorar a saúde pública global, preservando a eficácia dos poucos antibióticos disponíveis para combater as doenças infeciosas.
1. Nesme J, Cécillon S, Delmont TO, et al. Large-scale metagenomic-based study of antibiotic resistance in the environment. Curr Biol 2014; 24: 1096-100.
2. D’Costa VM, King CE, Kalan L, et al. Antibiotic resistance is ancient. Nature 2011; 477: 457-61.
3. Renwick MJ, Brogan DM, Mossialos E. A systematic review and critical assessment of incentive strategies for discovery and development of novel antibiotics. J Antibiot 2016; 69: 73-88.
4. Wright GD. The antibiotic resistome: the nexus of chemical and genetic diversity. Nat Rev Microbiol 2007; 5: 175-86.
5. Benveniste R, Davies J. Aminoglycoside antibiotic-inactivating enzymes in actinomycetes similar to those present in clinical isolates of antibiotic-resistant bacteria. Proc Natl Acad Sci USA 1973; 70: 2276-80.
6. Shoemaker NB, Vlamakis H, Hayes K, et al. Evidence for extensive resistance gene transfer among Bacteroides spp. and among Bacteroides and other genera in the human colon. Appl Environ Microbiol 2001; 67: 561-8.
7. Martínez JL, Coque TM, Baquero F. Prioritizing risks of antibiotic resistance genes in all metagenomes. Nat Rev Microbiol 2015; 13: 396.
8. Baker-Austin C, Wright MS, Stepanauskas R. Co-selection of antibiotic and metal resistance. Trends Microbiol 2006; 14: 176-82.
9. Wencewicz TA. Crossroads of Antibiotic Resistance and Biosynthesis. J Mol Biol 2019; 431: 3370-99.
10. Knapp CW, Dolfing J, Ehlert PA, et al. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940. Environ Sci Technol 2010; 44: 580-7.
11. Wolters B, Hauschild K, Blau K, et al. Biosolids for safe land application: does wastewater treatment plant size matters when considering antibiotics, pollutants, microbiome, mobile genetic elements and associated resistance genes? Environ Microbiol 2022; 24: 1573-89.
12. Dogra S, Sakwinska O, Soh SE, et al. Rate of establishing the gut microbiota in infancy has consequences for future health. Gut Microbes 2015; 6: 321-5.
13. Cox LM, Yamanishi S, Sohn J, et al. Altering the intestinal microbiota during a critical developmental window has lasting metabolic consequences. Cell 2014; 158: 705-21.
14. Li X, Stokholm J, Brejnrod A, et al. The infant gut resistome associates with E. coli, environmental exposures, gut microbiome maturity, and asthma-associated bacterial composition. Cell Host Microbe 2021; 29: 975-87.e4.
15. Mulder I, Siemens J, Sentek V, et al. Quaternary ammonium compounds in soil: implications for antibiotic resistance development. Rev Environ Sci Bio/Technology 2018; 17: 159-85.
16. Subramanian S, Huq S, Yatsunenko T, et al. Persistent gut microbiota immaturity in malnourished Bangladeshi children. Nature 2014; 510: 417-21.

