Microbioma intestinal y resistencia a los antimicrobianos
Por el Dr. Joseph Nesme
Profesor adjunto, Sección de Microbiología, Departamento de Biología, Universidad de Copenhague, Dinamarca
Espacio para el público general
Encuentra aquí tu espacio dedicado
en_sources_title
en_sources_text_start en_sources_text_end
Capítulos
Acerca de este artículo
La resistencia a los antibióticos es antigua y está extendida, y el resistoma ambiental es un reservorio de genes de resistencia que agrava la pandemia silenciosa de resistencia a los antimicrobianos. La transferencia genética horizontal, la dinámica de poblaciones y la coselección aceleran la aparición y la diseminación del resistoma, de modo que para el 2050, más de 10 millones de muertes al año estarán causadas por la resistencia a los antibióticos. La colonización temprana del microbioma intestinal puede conducir a disbiosis que desemboquen en una mayor abundancia y diversidad del resistoma intestinal de los lactantes. Adoptar el enfoque “Una sola salud”, es decir, tener en cuenta la interconexión de la salud humana, la salud animal y el medioambiente es fundamental para hacer frente a los complejos retos que plantea la resistencia a los antibióticos.
La resistencia a los antibióticos en el medioambiente: Un reto preexistente
La resistencia a los antibióticos (RA) es un fenómeno antiguo y extendido en el medioambiente, que existe desde mucho antes de la introducción de moléculas antibióticas como tratamiento. El medioambiente es un amplio reservorio de genes de resistencia a los antibióticos con comunidades microbianas diversas que albergan mecanismos de resistencia. Se ha observado RA en numerosos entornos ambientales, como el suelo, el agua, las plantas, los animales e incluso en el permafrost ártico de 30 000 años de antigüedad 1, 2 . La función ecológica de las moléculas antibióticas y la resistencia asociada en entornos no clínicos sigue sin estar clara, pero pone de relieve el hecho de que un conjunto de genes fácilmente disponible es anterior al uso clínico de los antibióticos, y explica la rápida aparición de patógenos. La actual crisis de los antibióticos es un fenómeno evolutivo, y las estrategias de mitigación deben tener en cuenta la ecología microbiana. Lo problemático es la rápida adquisición de resistencia por parte de patógenos que antes eran sensibles a los antibióticos, lo que lleva a los fracasos terapéuticos, sobre todo cuando está previsto que solo se comercialicen unos pocos antimicrobianos nuevos en el futuro 3 .
Mecanismos que fomentan la aparición y la diseminación del resistoma
El resistoma se refiere a la totalidad de genes que codifican las proteínas relacionadas con la resistencia a los antibióticos (RA) o las proteínas que tienen la capacidad de convertirse en agentes potentes de RA 4 (figura 1). Esto incluye genes de RA reconocidos en bacterias patogénicas (las problemáticas), genes de RA de organismos que producen antibióticos, como Streptomyces spp. que produce el antibiótico estreptomicina y el gen de resistencia asociado 5 , genes de RA crípticos (es decir, genes que podrían ofrecer resistencia en un entorno genético diferente, por ejemplo, bombas de eflujo reguladas al alza o porinas reguladas a la baja), y genes precursores de la RA que codifican proteínas con un nivel mínimo de afinidad o resistencia a los compuestos antibióticos.
Hay que destacar que una parte considerable de estos subconjuntos de genes se solapa debido a la homología de las secuencias, lo que indica antecedentes evolutivos comunes.
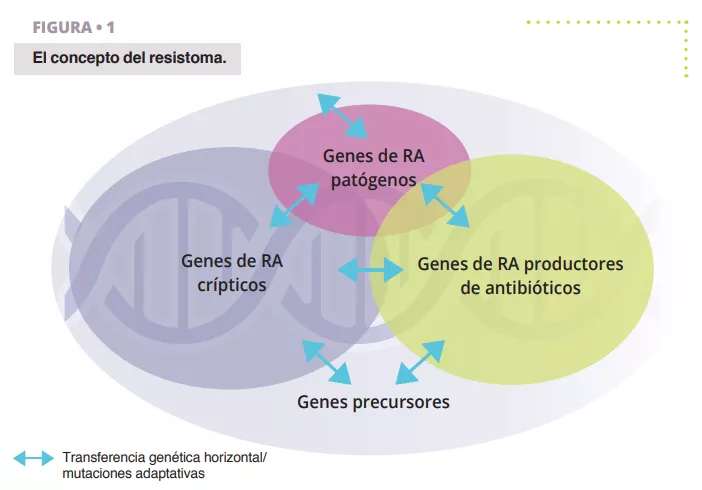
Es importante señalar que la noción de resistoma es diferente de una “RA funcional y clínicamente relevante”. Los genes del resistoma pueden pasar por los distintos estados descritos más arriba mediante transferencia genética horizontal (TGH), mutaciones puntuales o recombinación, lo que da lugar a nuevos anfitriones o contextos genéticos en los que puede expresarse el fenotipo de RA clínicamente relevante. Por lo tanto, un gen de resistencia no es problemático en sí, ya que depende del anfitrión y del contexto genético, pero todos los genes del resistoma representan una amenaza potencial con distintos riesgos asociados para la salud pública. El descubrimiento de un gen de resistencia a una molécula clínicamente relevante, localizado en un elemento móvil y alojado en un patógeno humano es un riesgo crítico, pero el mismo gen, o sus homólogos cercanos, encontrados en una bacteria del suelo no patógena y no asociado a un elemento genético móvil es un gen de RA de muy bajo riesgo. Por lo tanto, es fundamental clasificar el riesgo de resistencia a los antibióticos en los estudios sobre el resistoma.
La transferencia genética horizontal (TGH) es un mecanismo clave responsable de la rápida propagación de genes de RA entre las bacterias, incluso entre especies lejanas. Por ejemplo, Bacteroides spp., un grupo predominante en la microbiota intestinal, posee un gen de resistencia a los macrólidos ermB idéntico a los que se encuentran en numerosos Clostridium perfringens, Streptococcus pneumoniae y Enterococcus faecalis aislados de distintos orígenes, que indican la conexión genética entre Bacteroides y algunas bacterias grampositivas que no son predominantes en el intestino humano 6. Los elementos genéticos, como los plásmidos, facilitan la transferencia de genes de resistencia entre distintas especies microbianas 7. La TGH posibilita la diseminación de genes a través de distintos entornos y poblaciones bacterianas, lo que contribuye a la prevalencia y a la diversidad general de la RA. La coselección es otro factor importante en la propagación de la RA. El uso de compuestos no antibióticos, como metales pesados y biocidas, puede coseleccionar genes de RA ejerciendo presiones selectivas sobre las poblaciones microbianas, ya sea por corresistencia (distintos factores determinantes de resistencia presentes en el mismo elemento genético) o resistencia cruzada (el mismo factor determinante genético responsable de la resistencia a los antibióticos y a los metales) 8. La exposición a compuestos antimicrobianos de origen natural, como los producidos por microorganismos competidores o cualquier compuesto coselectivo, puede impulsar la selección de cepas resistentes 9. La presencia de antibióticos en el medioambiente, ya sean procedentes de fuentes naturales o de la actividad humana, contribuye aún más a la presión de selección de las resistencias. Además, el uso de antibióticos en la agricultura y en las prácticas veterinarias pueden conducir a la contaminación del medioambiente, lo que, con el tiempo, favorece la aparición y la propagación de genes de resistencia ambiental a los antibióticos 10.
Comprender las barreras y las tendencias de las transferencias de vectores genéticos que albergan genes de resistencia es fundamental para diseñar estrategias de mitigación ecoevolutivas e informadas para limitar la diseminación de la RA en los primeros años de vida y, en general, en contextos clínicos.
Disbiosis y resistoma intestinal en lactantes: Un equilibrio delicado
La diversidad del reservorio ambiental de genes de RA y su potencial para ser transferidos representa una amenaza para el microbioma intestinal humano en los primeros años de vida. Estrategias como un mejor tratamiento de las aguas residuales, un uso responsable de los antibióticos en la agricultura y en la medicina veterinaria, y una reducción de la contaminación ambiental con residuos antibióticos y bacterias resistentes a los antibióticos pueden ayudar a mitigar la propagación de la resistencia 11. Además, el seguimiento y la vigilancia de los reservorios ambientales puede proporcionar información valiosa sobre la aparición y la persistencia de la resistencia a los antibióticos para adaptar las intervenciones de salud pública.
Adoptar el enfoque “Una sola salud”, tener en cuenta la interconexión de la salud humana, la salud animal y el medioambiente es fundamental para hacer frente a los complejos retos que plantea la RA.
Después de nacer, el intestino es colonizado rápidamente por microorganismos adquiridos de la madre de su entorno. Durante los primeros años de vida es cuando se producen cambios drásticos, caracterizados por una baja resistencia, en comparación con el microbioma intestinal más estable de un adulto sano. Por lo tanto, los recién nacidos y los lactantes son más propensos a alteraciones en las comunidades microbianas, lo que se conoce como disbiosis. Durante ese período, hay numerosos factores que pueden influir y perturbar la maduración intestinal y, potencialmente, tener consecuencias a largo plazo sobre la salud 12. Estudios con ratones han demostrado que, durante este intervalo decisivo para el desarrollo, en lugar de un efecto directo de las moléculas antibióticas, es la alteración de la composición de la microbiota intestinal la que desencadena consecuencias metabólicas, como la obesidad 13 .
Descifrar el resistoma antibiótico en el intestino de lactantes: Conclusiones de un extenso estudio de cohortes
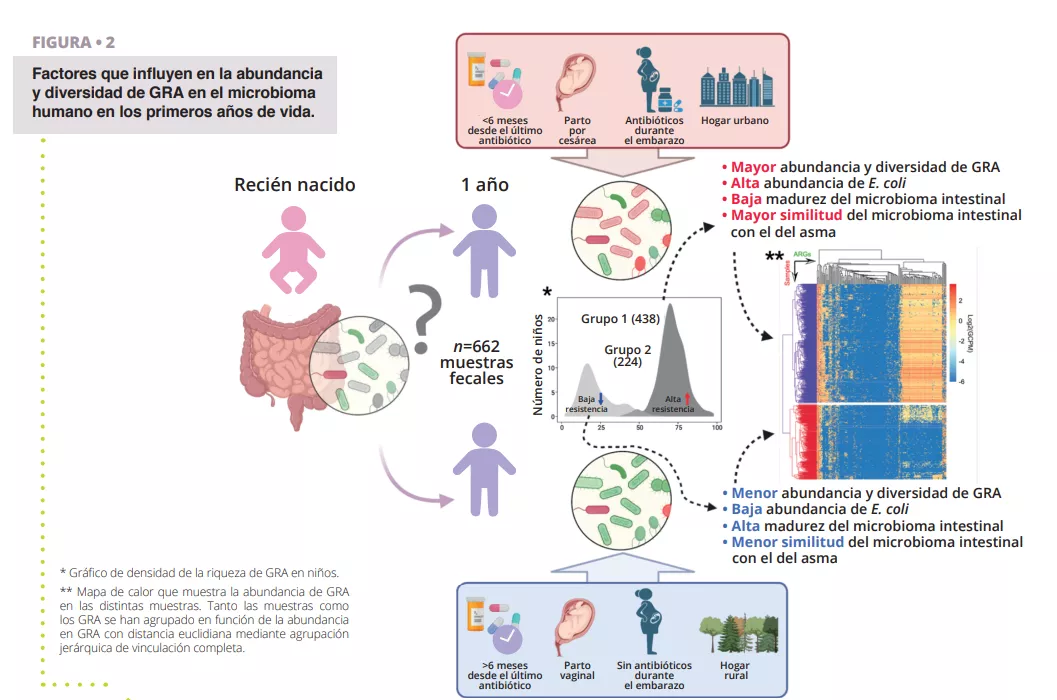
Aunque la resistencia a los antibióticos es problemática a cualquier edad, el establecimiento del microbioma intestinal en una edad temprana representa un intervalo de oportunidad para limitar el desarrollo de un reservorio de genes de RA en el intestino. Por lo tanto, es importante identificar los distintos factores que aumentan o reducen la abundancia de genes de RA que podrían propagarse a patógenos infecciosos y provocar el fracaso de tratamientos con antibióticos a lo largo de la vida. Para estudiar el resistoma global del intestino humano, los investigadores se sirven de planteamientos holísticos que interrogan tanto la presencia de especies como el potencial funcional de los genomas, incluido el resistoma bacteriano. Los investigadores utilizan la extracción de ADN ambiental a partir de muestras indirectas (como muestras de heces para el intestino) seguida de una secuenciación masiva no dirigida (metagenómica indiscriminada). Aproximadamente el 80 % de las especies de bacterias del intestino humano detectadas con herramientas moleculares no se pueden cultivar, en concreto, los anaerobios especializados que habitan en el intestino. Es probable que haya una gran cantidad de microorganismos organizados en agregados celulares de múltiples especies con codependencias metabólicas cuyo aislamiento puro de cepa sea difícil, si no imposible. Sin embargo, con métodos computacionales, es posible reconstruir genomas casi completos a partir de metagenomas y asociar los genes codificados a especies o incluso cepas concretas de bacterias. En un estudio reciente, los investigadores analizaron muestras fecales de 662 lactantes de una cohorte que hizo un seguimiento de los niños desde el nacimiento hasta los 7 años 14. Los objetivos del estudio eran establecer una visión general, a escala de cohorte, del resistoma al año e identificar los factores perinatales y ambientales asociados con la abundancia y la diversidad de GRA. Los investigadores realizaron una secuenciación metagenómica indiscriminada de muestras obtenidas al año de los 662 niños para identificar los GRA y los taxones bacterianos presentes en las muestras (figura 2). Aprovechando el extenso conjunto de datos, los autores pudieron reconstruir los genomas asociados a los metagenomas (GAM), lo que les permitió anotar con seguridad la taxonomía de los genomas recuperados y su contenido en genes de RA.
Uno de los primeros resultados observados fue que todos los niños tenían como mínimo un tipo de GRA a múltiples fármacos en el intestino, lo que indica que, incluso sin haber recibido un tratamiento antibiótico, existe un resistoma residente asociado al microbioma intestinal. De hecho, muchos genes de resistencia a múltiples fármacos se identificaron como bombas de eflujo. Estas proteínas son componentes normales de cualquier célula bacteriana, pero algunas pueden conferir resistencia a los antibióticos y son coseleccionadas con mucha facilidad por resistencia cruzada, por ejemplo, hacia metales pesados o biocidas, lo que posiblemente explica su gran abundancia en el intestino, pero también en todos los ambientes [8, 15]. Otro resultado sorprendente fue la clara división de la cohorte en dos grupos, en función del perfil de su resistoma. El primer grupo se caracterizó por una mayor diversidad de GRA y una abundancia relativa, con Escherichia coli como el mayor contribuidor de GRA, como se muestra en la figura 2. Esto concuerda con observaciones anteriores según las cuales Enterobacteriaceae son abundantes en los primeros años de vida, pero deberían disminuir rápidamente cuando la población de Bacteroidetes empieza a colonizar el intestino. En algunos niños, la alteración de esta maduración se puede asociar a la conjunción de varios factores, como el consumo de antibióticos, el tipo de parto, haberse criado en un entorno rural o urbano, factores que pueden retrasar la reducción de la población de Escherichia coli y conducir a un aumento del resistoma al año. Esto también se confirma por la observación de que una mayor abundancia de GRA se asocia con una menor puntuación de madurez del microbioma intestinal, basada en ratios de taxones específicos relacionados con la edad 16 .
Obviamente, el tratamiento con antibióticos durante el embarazo y el primer año de vida tuvo un impacto significativo en la abundancia del resistoma intestinal del lactante. Se pudo observar una clara tendencia temporal entre el tiempo (más de 6 meses o menos) desde la última toma de antibiótico de un niño y la abundancia de GRA en el intestino.
Esto indica una capacidad de recuperación a una edad temprana, que quizá se podría mejorar con una intervención dirigida, por ejemplo, con probióticos o prebióticos, y que aún se debe probar. Sin embargo, a escala de cohorte, las mayores diferencias en la abundancia de GRA se encontraron en genes de resistencia a antibióticos que no se recetaron a los niños, lo que indica que factores perinatales y ambientales, además del tratamiento con antibióticos, también influyen en el resistoma intestinal. Otra observación que se desprende de este estudio que conecta el entorno circundante y su resistoma asociado, y el resistoma intestinal, fue que los niños cuyos hogares estaban en zonas urbanas tenían una carga significativamente mayor de GRA que los niños que vivían en zonas rurales. Hay un sinfín de posibles factores de confusión que podrían explicar esto, pero refuerza el hecho de que la contribución del entorno al desarrollo del microbioma en los primeros años de vida es extremadamente importante.
Se puede plantear la hipótesis de que la vida urbana se asocia a un menor contacto con el exterior y a una menor diversidad del microbioma, o que el tipo de vivienda de los entornos rurales (casas) o urbanos (apartamentos) repercute en el microbioma interior, como se ha observado en el polvo de las camas.
Conclusión
La resistencia a los antibióticos es antigua y está extendida, y el resistoma ambiental es un reservorio de posibles genes de resistencia clínica. Los mecanismos como la transferencia genética horizontal, la compleja dinámica de poblaciones y los efectos de la coselección se han identificado como aceleradores en la evolución y propagación del resistoma antibiótico, lo que ha hecho que las cepas de patógenos que antes eran sensibles al tratamiento antibiótico ahora sean resistentes. El microbioma intestinal humano y su fase de desarrollo inicial son propensos a la disbiosis y a la posible invasión, selección y coselección de bacterias que albergan genes de resistencia, lo que puede tener consecuencias para la salud a largo plazo. No está claro hasta qué punto estarán relacionados con el fracaso de futuros tratamientos con antibióticos. Se necesitan estudios multiómicos a largo plazo y de alta resolución temporal para identificar, en primer lugar, los patrones longitudinales de desarrollo del microbioma intestinal y, en segundo lugar, las vías más probables de transferencia de genes de resistencia a los antibióticos mediante TGH. El conjunto de estos resultados ofrecerá información para diseñar estrategias de prevención que permitan minimizar el desarrollo de la RA desde las primeras etapas de la vida y limitar la transferencia a patógenos problemáticos. Este esfuerzo es de la máxima importancia para reducir las consecuencias de la pandemia mundial de resistencia a los antimicrobianos y mejorar la salud pública mundial, así como para mantener la eficacia de los pocos antibióticos disponibles para luchar contra las enfermedades infecciosas.
1. Nesme J, Cécillon S, Delmont TO, et al. Large-scale metagenomic-based study of antibiotic resistance in the environment. Curr Biol 2014; 24: 1096-100.
2. D’Costa VM, King CE, Kalan L, et al. Antibiotic resistance is ancient. Nature 2011; 477: 457-61.
3. Renwick MJ, Brogan DM, Mossialos E. A systematic review and critical assessment of incentive strategies for discovery and development of novel antibiotics. J Antibiot 2016; 69: 73-88.
4. Wright GD. The antibiotic resistome: the nexus of chemical and genetic diversity. Nat Rev Microbiol 2007; 5: 175-86.
5. Benveniste R, Davies J. Aminoglycoside antibiotic-inactivating enzymes in actinomycetes similar to those present in clinical isolates of antibiotic-resistant bacteria. Proc Natl Acad Sci USA 1973; 70: 2276-80.
6. Shoemaker NB, Vlamakis H, Hayes K, et al. Evidence for extensive resistance gene transfer among Bacteroides spp. and among Bacteroides and other genera in the human colon. Appl Environ Microbiol 2001; 67: 561-8.
7. Martínez JL, Coque TM, Baquero F. Prioritizing risks of antibiotic resistance genes in all metagenomes. Nat Rev Microbiol 2015; 13: 396.
8. Baker-Austin C, Wright MS, Stepanauskas R. Co-selection of antibiotic and metal resistance. Trends Microbiol 2006; 14: 176-82.
9. Wencewicz TA. Crossroads of Antibiotic Resistance and Biosynthesis. J Mol Biol 2019; 431: 3370-99.
10. Knapp CW, Dolfing J, Ehlert PA, et al. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940. Environ Sci Technol 2010; 44: 580-7.
11. Wolters B, Hauschild K, Blau K, et al. Biosolids for safe land application: does wastewater treatment plant size matters when considering antibiotics, pollutants, microbiome, mobile genetic elements and associated resistance genes? Environ Microbiol 2022; 24: 1573-89.
12. Dogra S, Sakwinska O, Soh SE, et al. Rate of establishing the gut microbiota in infancy has consequences for future health. Gut Microbes 2015; 6: 321-5.
13. Cox LM, Yamanishi S, Sohn J, et al. Altering the intestinal microbiota during a critical developmental window has lasting metabolic consequences. Cell 2014; 158: 705-21.
14. Li X, Stokholm J, Brejnrod A, et al. The infant gut resistome associates with E. coli, environmental exposures, gut microbiome maturity, and asthma-associated bacterial composition. Cell Host Microbe 2021; 29: 975-87.e4.
15. Mulder I, Siemens J, Sentek V, et al. Quaternary ammonium compounds in soil: implications for antibiotic resistance development. Rev Environ Sci Bio/Technology 2018; 17: 159-85.
16. Subramanian S, Huq S, Yatsunenko T, et al. Persistent gut microbiota immaturity in malnourished Bangladeshi children. Nature 2014; 510: 417-21.

