Le microbiote intestinal et la résistance antimicrobienne
Par le Dr Joseph Nesme
Professeur assistant, Microbiologie, Département de biologie, Université de Copenhague, Danemark
Section grand public
Retrouvez ici votre espace dédié
en_sources_title
en_sources_text_start en_sources_text_end
Chapitres
A propos de cet article
La résistance aux antibiotiques est ancienne et répandue, et le résistome environnemental est un réservoir de gènes de résistance alimentant la pandémie silencieuse de la résistante antimicrobienne. Le transfert horizontal de gènes, les dynamiques démographiques et la co-sélection accélèrent l’émergence et la dissémination du résistome, et, en 2050, plus de 10 millions de décès seront causés chaque année par la résistance aux antibiotiques. La colonisation précoce du microbiote intestinal favorise la dysbiose, qui peut entraîner une abondance et une diversité accrues des gènes du résistome intestinal des nourrissons. L’adoption d’une approche « One Health » tenant compte de l’interconnexion, entre la santé humaine, la santé animale et l’environnement est essentielle pour résoudre les défis complexes posés par la résistance aux antibiotiques.
Résistance aux antibiotiques dans l’environnement : un problème préexistant
La résistance aux antibiotiques (RA) est un phénomène ancien et répandu dans l’environnement, qui existait déjà bien avant l’introduction des molécules antibiotiques en tant que traitement. L’environnement sert de vaste réservoir de gènes de résistance aux antibiotiques : diverses communautés microbiennes hébergent des mécanismes de résistance. Des gènes de RA ont été trouvés dans différents milieux environnementaux, dont la terre, l’eau, les plantes, les animaux, et même le permafrost arctique vieux de 30 000 ans [1, 2]. Le rôle écologique des molécules antibiotiques et de la résistance associée dans les environnements non cliniques reste incertain, mais souligne le fait qu’un groupe de gènes facilement accessibles précède l’utilisation clinique d’antibiotiques et explique leur émergence rapide chez les agents pathogènes. La crise actuelle des antibiotiques est un phénomène d’évolution, et les stratégies d’atténuation doivent prendre en compte l’écologie microbienne. Le problème émerge du développement rapide de la résistance par les agents pathogènes précédemment sensibles, qui entraîne l’échec des traitements. Cette problématique est exacerbée par le fait que très peu de nouveaux antimicrobiens sont attendus sur le marché [3].
Mécanismes favorisant l’émergence et la dissémination du résistome
Le résistome désigne l’ensemble des gènes qui encodent les protéines liées à la résistance aux antibiotiques (RA) ou les protéines pouvant potentiellement évoluer pour devenir des agents RA puissants [4] (figure 1). Cet ensemble inclue les gènes RA reconnus des bactéries pathogènes (celles qui posent problème), les gènes RA issus des organismes produisant des antibiotiques, comme les Streptomyces spp., qui produisent l’antibiotique streptomycine et le gène de résistance associé [5], les gènes RA cryptiques (c.-à-d. des gènes pouvant générer de la résistance dans un contexte génétique différent ; p. ex., des pompes d’efflux surexprimées ou des porines sous-exprimées), et les gènes RA précurseurs codant des protéines avec un niveau minimum d’affinité ou de résistance aux composés antibiotiques.
Il est à noter qu’une partie considérable de ces sous-groupes de gènes se recoupent avec des homologies séquentielles, indiquant qu’ils ont probablement une origine évolutive commune.
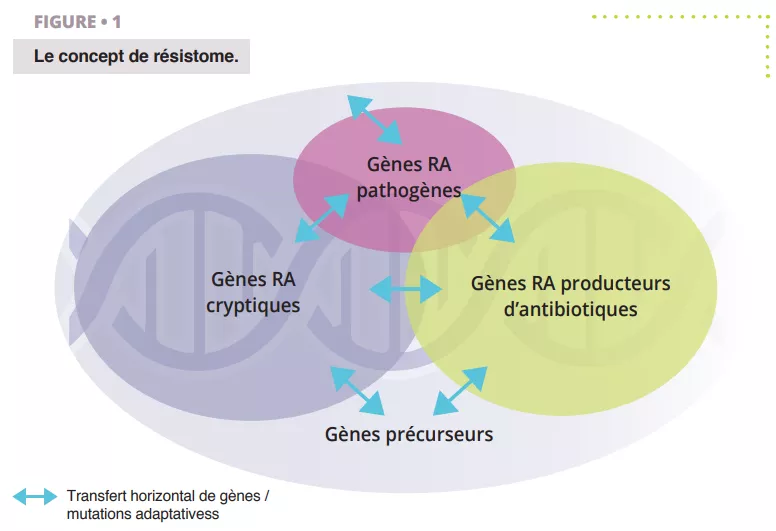
La notion de résistome est distincte de celle de « RA fonctionnelle et cliniquement pertinente ». En effet, les gènes du résistome peuvent transiter entre les différents états décrits ci-dessus via le transfert horizontal de gènes (THG), les mutations ponctuelles ou la recombinaison, qui conduisent à de nouveaux hôtes ou à un nouveau contexte génétique où les phénotypes RA cliniquement pertinents peuvent être exprimés. Un gène de résistance n’est donc pas problématique en tant que tel, car il est dépendant de l’hôte et du contexte génétique, mais tous les gènes du résistome représentent une menace potentielle et sont associés à différents risques pour la santé publique. La découverte d’un gène de résistance à une molécule cliniquement pertinente, situé sur un élément mobile et hébergé par un agent pathogène humain représente un risque critique, mais le même gène, ou ses homologues proches, trouvés dans une bactérie terricole non pathogène et non associée à l’élément génétique mobile, représentent des gènes RA à faible risque. Évaluer le risque associé à la résistance aux antibiotiques est donc primordial dans les études sur le résistome.
Le transfert horizontal de gènes (THG) est un mécanisme clé responsable de la propagation rapide de gènes RA parmi les bactéries, même entre des lignées lointaines. Par exemple, Bacteroides spp., un groupe prédominant dans le microbiote intestinal humain, possède le gène de résistance aux macrolides ermB, identique au gène trouvé dans plusieurs isolats de Clostridium perfringens, Streptococcus pneumoniae et Enterococcus faecalis de diverses origines géographiques, ce qui indique une connexion génétique entre les Bacteroides et certaines bactéries à Gram positif peu répandues dans l’intestin humain [6]. Des éléments génétiques comme les plasmides facilitent le transfert des gènes de résistance entre différentes espèces microbiennes [7]. Le THG permet la dissémination de gènes dans différents environnements et populations bactériennes, contribuant à la prévalence générale et à la diversité des gènes de RA. La co-sélection est un autre facteur significatif dans la propagation de la RA. L’utilisation de composés non antibiotiques, comme les métaux lourds et les biocides, peut permettre la co-sélection des gènes RA en exerçant des pressions sélectives sur les populations microbiennes, soit par co-résistance (différents déterminants de la résistance présents sur le même élément génétique), soit par résistance croisée (le même déterminant génétique est responsable de la résistance aux antibiotiques et aux métaux) [8]. L’exposition aux composés antimicrobiens naturellement présents, comme ceux produits par les micro-organismes compétiteurs ou tout composé co-sélectif, peut favoriser la sélection de souches résistantes [9]. La présence d’antibiotiques dans l’environnement, issus soit de sources naturelles, soit d’activités humaines, contribue également à la pression sélective pour la résistance. Par ailleurs, l’utilisation d’antibiotiques en agriculture et dans les pratiques vétérinaires peut entraîner une contamination de l’environnement et ainsi favoriser au fil du temps l’émergence et la propagation des gènes de résistance environnementale aux antibiotiques [10].
Comprendre les obstacles et les tendances qui régulent le transfert de vecteurs génétiques hébergeant des gènes de résistance est crucial pour élaborer des stratégies d’atténuation éco-évolutives éclairées, afin de limiter la dissémination de la RA en début de vie et, de façon générale, dans les environnements cliniques.
La dysbiose et le résistome intestinal chez les nourrissons : un équilibre délicat
La diversité du réservoir environnemental de gènes RA et sa capacité d’être potentiellement transféré représentent une menace pour le microbiote intestinal humain en début de vie. Les stratégies comme le traitement amélioré des eaux usées, l’usage responsable d’antibiotiques en agriculture et en médecine vétérinaire, ainsi que la réduction de la contamination environnementale avec des résidus de bactéries résistantes aux antibiotiques peut contribuer à limiter la propagation de la résistance [11]. De plus, le contrôle et la surveillance des réservoirs environnementaux peuvent fournir de précieuses informations sur l’émergence et la persistance des gènes RA et éclairer les interventions liées à la santé publique.
L’adoption d’une approche « One Health » tenant compte de l’interconnexion, entre la santé humaine, la santé animale et l’environnement est essentielle pour résoudre les défis complexes posés par la résistance aux antibiotiques.
Après la naissance, notre intestin est rapidement colonisé par des micro-organismes issus de la mère et de l’environnement proche. C’est pendant les premières années de vie que les changements sont drastiques et caractérisés par une résilience faible, en comparaison du microbiote intestinal plus stable d’un adulte sain. Les nouveau-nés et les nourrissons sont donc plus sujets aux perturbations des communautés microbiennes, appelées dysbioses. Pendant cette période, de nombreux facteurs peuvent influencer et perturber la maturation intestinale, et potentiellement entraîner des conséquences à long terme sur la santé [12]. Des études sur des souris ont montré que pendant cette fenêtre de développement critique, c’est l’altération de la composition du microbiote intestinal, plutôt que l’effet direct des molécules antibiotiques, qui entraîne des conséquences métaboliques comme l’obésité [13].
Analyse du résistome intestinal des nourrissons : informations issues d’une vaste étude de cohorte
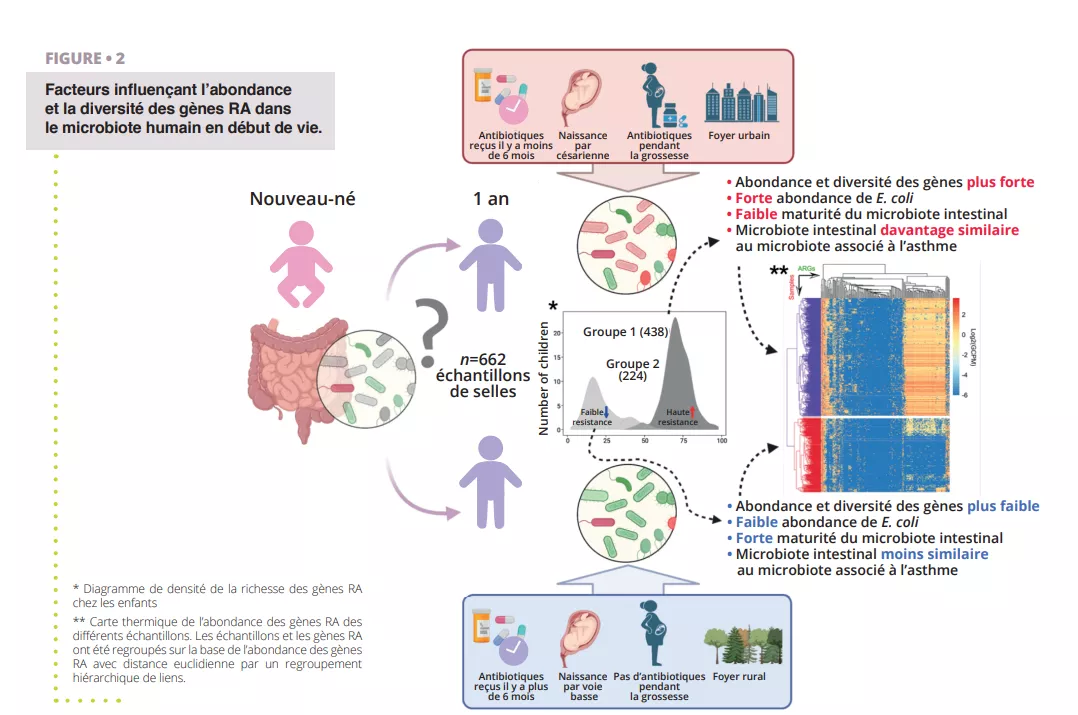
Bien que la résistance aux antibiotiques soit problématique à tous les âges de la vie, la formation du microbiote intestinal à un âge précoce représente une fenêtre d’opportunité pour limiter l’accumulation de gènes RA dans l’intestin. Il est donc important d’identifier les différents facteurs qui augmentent ou réduisent l’abondance des gènes RA pouvant se propager à des agents pathogènes infectieux et provoquer l’échec des traitements antibiotiques au cours de la vie. Pour étudier le résistome intestinal humain dans son ensemble, les chercheurs s’appuient sur une approche générale se penchant à la fois sur la présence des différentes espèces et le potentiel fonctionnel des génomes, qui inclut le résistome. Les chercheurs s’appuient sur l’extraction d’ADN environnemental d’échantillons équivalents (p. ex., échantillon de selles pour les intestins), suivie par le séquençage haut débit non ciblé (séquençage shotgun métagénomique). Environ 80 % des espèces de bactéries de l’intestin humain détectées par les outils moléculaires ne sont pas cultivables, en particulier les anaérobies spécialisés présents dans l’intestin. Il est probable que de nombreux microorganismes soient organisés en agrégats de cellules multi-espèces avec des co-dépendances métaboliques, ce qui rend l’isolement de souches pures délicat, voire impossible. Grâce à des méthodes informatiques, il est cependant possible de reconstruire des génomes quasi complets de métagénomes et de gènes encodés associés à partir d’espèces spécifiques ou de souches de bactéries. Dans une étude récente, des chercheurs ont analysé des échantillons de matière fécale de 662 nourrissons provenant d’une cohorte d’enfants suivis depuis la naissance jusqu’à l’âge de 7 ans [14]. Les objectifs de l’étude étaient de créer un aperçu, à l’échelle d’une cohorte, du résistome à l’âge de 1 an et d’identifier les facteurs périnataux et environnementaux associés à l’abondance et à la diversité des gènes AR. Les chercheurs ont utilisé le séquençage shotgun métagénomique d’échantillons obtenus à l’âge de 1 an issus de 662 enfants pour identifier les gènes RA et les taxons bactériens présents dans les échantillons (figure 2). Utilisant leur vaste ensemble de données, les auteurs ont été capables de reconstruire les génomes assemblés à partir de données métagénomiques (MAG), ce qui leur a permis de déterminer avec assurance la taxinomie des génomes reconstruits et leur contenu génétique RA.
L’un des premiers résultats observés était que les nourrissons avaient au moins un type de gène AR résistant à plusieurs médicaments dans leur intestin, ce qui indique que même en l’absence de traitement antibiotique, il existe un résistome fixe associé au microbiote intestinal. En effet, de nombreux gènes de résistance à plusieurs médicaments ont été identifiés comme des pompes d’efflux. Ces protéines sont des composants normaux de chaque cellule bactérienne, mais certaines peuvent engendrer une résistance aux antibiotiques et sont très facilement sélectionnées par résistance croisée, par exemple aux métaux lourds ou aux biocides, ce qui explique potentiellement leur forte abondance dans l’intestin, mais également dans tous les environnements [8, 15]. Un autre résultat frappant était la nette séparation de la cohorte en deux groupes, selon leur profil de résistome. Le premier groupe était caractérisé par une diversité et une abondance relative plus élevées de gènes RA, avec Escherichia coli en tant que principal contributeur des gènes RA, comme indiqué dans la figure 2. Cette constatation s’accorde avec les observations antérieures, selon lesquelles les Enterobacteriaceae sont abondantes en début de vie, mais qu’elles doivent diminuer rapidement lorsque les populations de Bacteroidetes commencent à coloniser l’intestin. L’altération de cette maturation chez certains enfants peut être associée à une combinaison de plusieurs facteurs, comme l’utilisation d’antibiotiques, le type d’accouchement ou le caractère rural ou urbain du foyer, qui semblent retarder la réduction de la population d’Escherichia coli et favoriser la présence d’un résistome accru à l’âge de 1 an. Cette altération est également confirmée par l’observation selon laquelle l’abondance plus élevée de gènes RA est associée à un score de maturité du microbiote intestinal plus faible, sur la base de ratios entre des taxons spécifiques liés à l’âge [16].
Le traitement antibiotique pendant la grossesse et la première année du nourrisson avait évidemment un impact sur l’abondance du résistome intestinal de l’enfant. Une tendance temporelle nette a pu être observée entre le temps passé (supérieur ou inférieur à 6 mois) depuis le dernier traitement antibiotique pris par un enfant et l’abondance de gènes RA dans son intestin.
Cette donnée indique un certain niveau de résilience à un âge précoce, qui pourrait potentiellement être améliorée par une intervention ciblée, p. ex., avec des probiotiques ou des prébiotiques, et reste à tester. À l’échelle de la cohorte, les plus grandes différences en matière d’abondance de gènes RA ont cependant été trouvées dans les gènes de résistance à des antibiotiques qui n’avaient pas été prescrits aux enfants, ce qui implique que les facteurs périnataux et environnementaux, en dehors du traitement antibiotique, influencent également le résistome intestinal. Autre observation de cette étude en lien avec l’environnement proche et le résistome intestinal associé : les enfants dont le foyer se trouvait dans des zones urbaines présentaient une quantité significativement plus élevée de gènes RA que les enfants vivant dans des zones rurales. Il existe une multitude de facteurs de confusion pouvant expliquer cette différence, mais cette observation renforce l’idée que la contribution de l’environnement au développement du microbiote en début de vie est extrêmement importante.
Parmi les différentes hypothèses, on peut penser que la vie urbaine implique moins de contact avec l’extérieur et une réduction de la diversité du microbiote, ou que le type de logement associé aux environnements ruraux (maison) ou urbains (appartement) a des conséquences sur le microbiote en intérieur, comme on peut le voir dans la composition de la poussière de lit.
Conclusion
La résistance aux antibiotiques est ancienne et répandue, et le résistome environnemental est un réservoir de gènes de résistance clinique potentiels. Des mécanismes tels que le transfert horizontal de gènes, les dynamiques démographiques complexes et les effets de co-sélection ont été identifiés comme des facteurs significatifs d’accélération de l’évolution et de la propagation du résistome antibiotique, qui rendent résistantes au traitement antibiotique les souches d’agents pathogènes précédemment sensibles aux antibiotiques. Le microbiome intestinal humain et sa phase de développement précoce sont sujets à la dysbiose et à l’invasion, sélection et co-sélection potentielles de bactéries hébergeant des gènes de résistance, qui peuvent avoir des conséquences à long terme sur la santé. La nature de la relation de ce phénomène avec l’échec de futurs traitements antibiotiques reste incertaine. Une haute résolution temporelle et des études multiomiques à long terme sont nécessaires pour identifier, en premier lieu, les tendances longitudinales du développement du microbiome intestinal et, dans un second temps, les voies les plus probables pour le transfert de gènes RA par THG. Ensemble, ces résultats fourniront des informations permettant d’élaborer des stratégies préventives pour atténuer le développement de la RA dès un stade précoce de la vie et limiter son transfert vers des agents pathogènes problématiques. Cette démarche est de la plus haute importance pour réduire le fardeau de la pandémie mondiale de résistance aux antimicrobiens et améliorer la santé publique à travers le monde en préservant l’efficacité des quelques antibiotiques disponibles pour lutter contre les maladies infectieuses.
1. Nesme J, Cécillon S, Delmont TO, et al. Large-scale metagenomic-based study of antibiotic resistance in the environment. Curr Biol 2014; 24: 1096-100.
2. D’Costa VM, King CE, Kalan L, et al. Antibiotic resistance is ancient. Nature 2011; 477: 457-61.
3. Renwick MJ, Brogan DM, Mossialos E. A systematic review and critical assessment of incentive strategies for discovery and development of novel antibiotics. J Antibiot 2016; 69: 73-88.
4. Wright GD. The antibiotic resistome: the nexus of chemical and genetic diversity. Nat Rev Microbiol 2007; 5: 175-86.
5. Benveniste R, Davies J. Aminoglycoside antibiotic-inactivating enzymes in actinomycetes similar to those present in clinical isolates of antibiotic-resistant bacteria. Proc Natl Acad Sci USA 1973; 70: 2276-80.
6. Shoemaker NB, Vlamakis H, Hayes K, et al. Evidence for extensive resistance gene transfer among Bacteroides spp. and among Bacteroides and other genera in the human colon. Appl Environ Microbiol 2001; 67: 561-8.
7. Martínez JL, Coque TM, Baquero F. Prioritizing risks of antibiotic resistance genes in all metagenomes. Nat Rev Microbiol 2015; 13: 396.
8. Baker-Austin C, Wright MS, Stepanauskas R. Co-selection of antibiotic and metal resistance. Trends Microbiol 2006; 14: 176-82.
9. Wencewicz TA. Crossroads of Antibiotic Resistance and Biosynthesis. J Mol Biol 2019; 431: 3370-99.
10. Knapp CW, Dolfing J, Ehlert PA, et al. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940. Environ Sci Technol 2010; 44: 580-7.
11. Wolters B, Hauschild K, Blau K, et al. Biosolids for safe land application: does wastewater treatment plant size matters when considering antibiotics, pollutants, microbiome, mobile genetic elements and associated resistance genes? Environ Microbiol 2022; 24: 1573-89.
12. Dogra S, Sakwinska O, Soh SE, et al. Rate of establishing the gut microbiota in infancy has consequences for future health. Gut Microbes 2015; 6: 321-5.
13. Cox LM, Yamanishi S, Sohn J, et al. Altering the intestinal microbiota during a critical developmental window has lasting metabolic consequences. Cell 2014; 158: 705-21.
14. Li X, Stokholm J, Brejnrod A, et al. The infant gut resistome associates with E. coli, environmental exposures, gut microbiome maturity, and asthma-associated bacterial composition. Cell Host Microbe 2021; 29: 975-87.e4.
15. Mulder I, Siemens J, Sentek V, et al. Quaternary ammonium compounds in soil: implications for antibiotic resistance development. Rev Environ Sci Bio/Technology 2018; 17: 159-85.
16. Subramanian S, Huq S, Yatsunenko T, et al. Persistent gut microbiota immaturity in malnourished Bangladeshi children. Nature 2014; 510: 417-21.

