Microbiote intestinal et métabolisme des médicaments
Synthèse
Par le Pr. Emily P. Balskus
Département de Chimie et de Biologie chimique, Université de Harvard, Cambridge, États-Unis
Section grand public
Retrouvez ici votre espace dédié
en_sources_title
en_sources_text_start en_sources_text_end
Chapitres
A propos de cet article
Auteur
Le microbiote intestinal transforme les structures chimiques des substances ingérées, y compris les médicaments à petites molécules administrés par voie orale. Ce métabolisme, qui peut considérablement varier d’un patient à l’autre, a un impact à la fois positif et négatif sur l’efficacité des médicaments et il peut également influer sur leur toxicité. Son rôle potentiel dans la variabilité interindividuelle de la réponse aux médicaments est de plus en plus reconnu depuis ces 10 dernières années. Nous allons ici étudier ce phénomène en nous concentrant en particulier sur les récentes avancées réalisées et sur leur impact potentiel futur sur la prise en charge des patients et la découverte des médicaments.
Les milliers de milliards de micro-organismes qui peuplent l’intestin humain possèdent un nombre de gènes bien plus important que le génome de l’hôte. Beaucoup de ces gènes codent pour des catalyseurs protéiques (enzymes) qui permettent aux micro-organismes intestinaux d’opérer un large éventail de réactions chimiques venant enrichir la chimie associée au corps humain. L’une des caractéristiques principales du métabolisme microbien intestinal est sa variabilité : à l’image de la composition du microbiote, les capacités métaboliques de cette communauté diffèrent selon les individus. À mesure que se poursuit le travail d’identification des liens entre le microbiote intestinal et la santé, il devient de plus en plus important de caractériser les transformations métaboliques microbiennes au niveau moléculaire.
L’une des propriétés majeures du microbiote intestinal est sa capacité à modifier chimiquement les structures des médicaments à petites molécules [1]. Les médicaments administrés par voie orale peuvent rencontrer les micro-organismes intestinaux soit avant d’être absorbés dans l’intestin grêle, soit dans le côlon si leur biodisponibilité orale est faible. Les médicaments administrés par voie orale ou injectés, ou leurs métabolites, entrent également en contact avec le microbiote s’ils sont excrétés par voie biliaire dans l’intestin. Dans la mesure où l’activité pharmacologique d’un médicament est directement liée à sa structure chimique, le métabolisme microbien peut avoir un effet important sur son action.
Effets de la métabolisation des médicaments par le microbiote intestinal
Le métabolisme microbien intestinal produit plusieurs conséquences en aval sur l’action et l’efficacité des médicaments (Figure 1). Comme le montrent les exemples des médicaments azoïques, la métabolisation de « promédicaments » (précurseurs inactifs) par des micro-organismes pourrait être nécessaire pour générer l’agent pharmacologique actif. La connaissance de ce phénomène est à l’origine de la conception de nouvelles stratégies basées sur les activités métaboliques microbiennes visant à libérer les médicaments de manière ciblée dans le côlon. La métabolisation assurée par le microbiote intestinal peut également avoir des effets négatifs sur l’activité des médicaments en perturbant les interactions avec les cibles prévues chez l’hôte. Prenons l’exemple de la digoxine, une molécule naturelle utilisée pour traiter certaines affections cardiaques. Chez 5 à 10 % des patients, le microbiote intestinal réduit le cycle lactone αβ-insaturé de la digoxine pour donner la dihydrodigoxine. Cette modification subtile opérée par la bactérie intestinale Eggerthella lenta réduit fortement l’affinité de liaison de la digoxine pour sa cible (la Na+/K+ ATPase) et entraîne donc une perte d’efficacité [2]. Un autre exemple est celui du principal médicament utilisé pour traiter la maladie de Parkinson, la L-dopa. La métabolisation de la L-dopa en dopamine par des enzymes de l’hôte au niveau du cerveau est essentielle pour le soulagement des symptômes. La métabolisation de la L-dopa par le microbiote intestinal produit également de la dopamine [3,4]. Étant donné que la dopamine générée au niveau périphérique ne peut pas franchir la barrière hémato-encéphalique, cette activité pourrait réduire la quantité de L-dopa atteignant le cerveau.
Pour finir, en plus de réduire l’activité, les modifications chimiques opérées par les micro-organismes intestinaux peuvent également produire une toxicité non voulue. Par exemple, le métabolisme microbien intestinal a été impliqué dans la létalité associée à la co-administration du médicament antiviral sorivudine et d’agents chimiothérapeutiques de type fluoropyrimidine. Il s’est avéré que le mécanisme en cause était la métabolisation, par le microbiote intestinal, de la sorivudine en bromovinyluracile. Ce métabolite inhibe en effet une enzyme clé de l’hôte impliquée dans la détoxification du 5-fluorouracile, ce qui augmente sa concentration à des niveaux létaux.
Les études de la métabolisation des médicaments par le microbiote intestinal ont débuté il y a plus de 80 ans quand on a découvert que l’antibiotique Prontosil, une molécule azoïque inactive vis-à-vis des isolats bactériens mais montrant une efficacité in vivo, subissait une réduction sous l’action du microbiote intestinal pour donner l’agent actif sulfanilamide.
D’autres exemples ont été découverts depuis, souvent en lien avec l’observation d’une variabilité au niveau de l’efficacité ou de la toxicité chez les patients. Pourtant, malgré ces découvertes, ces activités ne sont généralement toujours pas prises en compte dans le développement ou l’administration des médicaments.
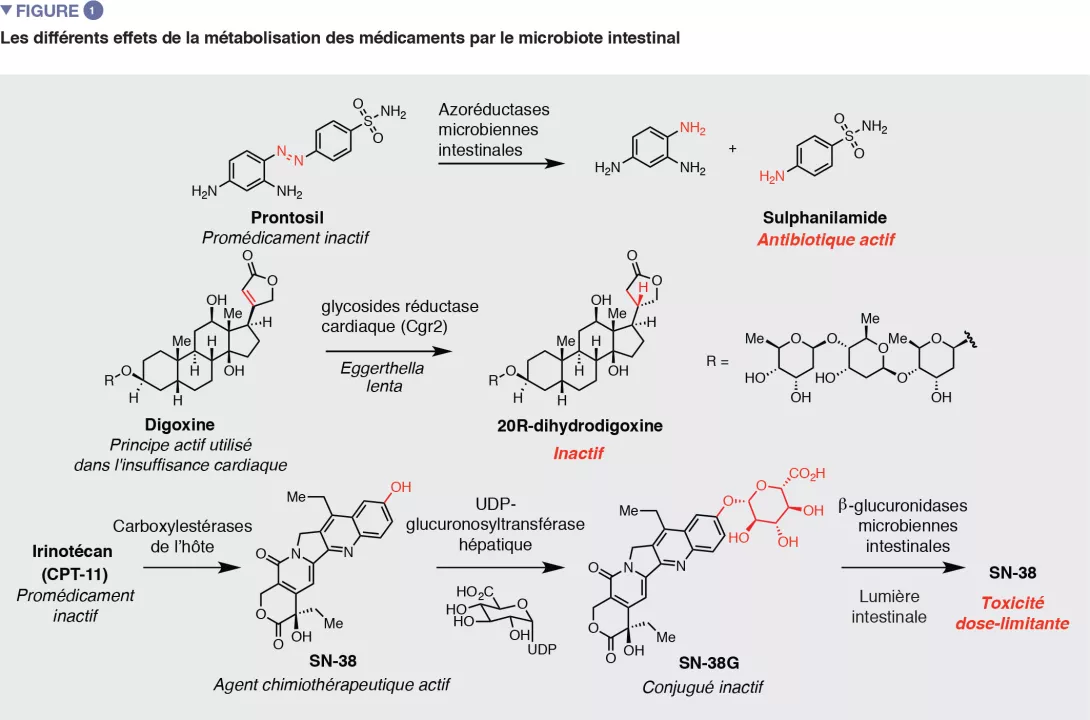
Effets du microbiote intestinal sur le métabolisme des médicaments : certaines bactéries intestinales peuvent activer ou inactiver des substances pharmaceutiques comme la digoxine, le prontosil ou l’irinotécan. Ces transformations modulent l’efficacité et la toxicité des traitements, illustrant le rôle central du microbiote dans la pharmacocinétique.
Une caractéristique importante de la métabolisation des médicaments par le microbiote intestinal est sa variabilité entre les patients, dont l’origine est la variabilité du microbiote intestinal. Même si certaines activités métaboliques sont retrouvées dans de nombreux micro-organismes, d’autres sont assurées par un petit sous-groupe peu abondant de la communauté intestinale. Le métabolisme peut varier entre des souches de la même espèce, et des bactéries même étroitement liées peuvent présenter des différences importantes au niveau de leur génome. Il n’est donc peut-être pas surprenant que la composition de la communauté soit souvent un mauvais facteur prédictif du métabolisme et que le métabolisme de certains médicaments puisse être rapide chez certaines personnes et absent chez d’autres. Cette variabilité a probablement des conséquences importantes mais incomplètement comprises pour les patients prenant différents médicaments à petites molécules.
Compréhension du métabolisme des médicaments au niveau moléculaire
Afin de bien comprendre la métabolisation des médicaments par le microbiote intestinal, il faut établir des liens entre les activités individuelles et les micro-organismes, les gènes et les enzymes. L’identification des micro-organismes métabolisant des médicaments spécifiques est typiquement nécessaire pour permettre de réaliser les études des mécanismes en aval. Pour ce faire, on peut cribler les isolats microbiens intestinaux disponibles ou isoler les micro-organismes métabolisateurs directement dans des échantillons de microbiote intestinal complexe. Il faut ensuite relier les transformations d’intérêt aux gènes et aux enzymes. Cette étape est cruciale pour étudier le métabolisme dans des communautés intestinales complexes car les gènes codant pour les enzymes métaboliques permettent de détecter et de prédire les activités individuelles dans les génomes microbiens et les données de séquençage du microbiome. Le lien entre le métabolisme des médicaments et les gènes microbiens peut être établi de différentes manières, notamment par l’analyse rationnelle des génomes à la recherche d’enzymes possédant les capacités catalytiques requises, l’utilisation du séquençage de l’ARN pour identifier les gènes dont l’expression est spécifiquement augmentée en réponse à un médicament, et l’utilisation de la génomique comparative pour associer les gènes à des capacités métaboliques.
Identification de nouvelles activités métaboliques
En 2019, environ 60 exemples de métabolisation des médicaments par le microbiote intestinal avaient été rapportés. Deux récentes études ont utilisé des approches issues du criblage à haut débit et de l’expérimentation pour réaliser des recherches à grande échelle sur la métabolisation des médicaments par le microbiote intestinal, et ont ainsi fortement élargi l’éventail des transformations connues. Goodman et son équipe ont criblé 76 isolats bactériens intestinaux humains pour analyser leur capacité à métaboliser 271 médicaments à petites molécules et ils ont constaté que les deux tiers des médicaments étaient modifiés par au moins un micro-organisme [8]. Le groupe de Donia a réalisé un criblage analogue sur 575 médicaments en utilisant un échantillon de microbiote intestinal de patient ex vivo et il a découvert 45 nouvelles transformations [6]. Ces travaux suggèrent que les médicaments susceptibles d’être ainsi métabolisés pourraient être plus nombreux que ceux précédemment identifiés. Toutefois, la grande majorité de ces activités nouvellement rapportées n’ont pas encore été confirmées in vivo, donc on ne connaît pas leur signification pour les patients.
La chimie de la métabolisation des médicaments par le microbiote intestinal, qui a tendance à être réductrice et hydrolytique, est souvent distincte de celle des transformations
de l’hôte, qui impliquent une oxydation des médicaments et une conjugaison avec des métabolites plus polaires pour faciliter l’excrétion. Le métabolisme microbien a souvent des effets inverses sur la disponibilité des médicaments en prolongeant leur circulation dans l’organisme. Cependant, les transformations induites par le microbiote n’ont pas besoin d’être différentes pour avoir un impact sur l’action des médicaments. En effet, de récentes études réalisées avec l’antiviral brivudine suggèrent que ces activités pourraient affecter la pharmacocinétique du médicament même quand elles sont identiques au métabolisme de l’hôte [5].
Manipulation de la métabolisation des médicaments par le microbiote intestinal
Une fois que l’on sait que le microbiote intestinal transforme un médicament à petites molécules, la prochaine étape logique est de chercher à savoir comment cette activité peut être contrôlée, à la fois pour évaluer les conséquences du métabolisme sur l’action du médicament et pour améliorer le traitement du patient si ce métabolisme s’avère nocif. Différentes méthodes ont été utilisées à cette fin. Grâce aux modèles animaux gnotobiotiques (animaux axéniques colonisés de manière contrôlée avec un microbiote défini), on peut comparer des communautés contenant des souches intestinales métabolisant les médicaments ou des mutants de délétion dépourvus d’activités spécifiques. L’utilité de cette approche a été parfaitement illustrée par les études du laboratoire de Goodman sur la brivudine [5].
Cependant, la manipulation génétique est difficile dans les communautés microbiennes complexes natives et des approches alternatives sont donc évaluées. Une stratégie potentielle consiste à utiliser les connaissances en matière de physiologie bactérienne intestinale pour guider la manipulation de l’environnement intestinal via des interventions alimentaires. Pour la digoxine par exemple, Turnbaugh et son équipe ont observé que la présence de L-arginine diminue la métabolisation de la molécule par E. lenta [2]. Ils ont ensuite montré que l’administration d’aliments riches en protéines à des souris gnotobiotiques colonisées avec E. lenta réduisait l’inactivation de cette molécule in vivo.
Une autre stratégie très intéressante consiste à identifier des petites molécules qui inhibent l’activité d’enzymes microbiennes intestinales métabolisant les médicaments, comme l’a montré le laboratoire de Redinbo dans ses études sur le métabolisme de l’irinotécan. L’irinotécan est un promédicament qui est métabolisé par les cellules de l’hôte en SN-38, inhibiteur actif des topo-isomérases. Le SN-38 est métabolisé par l’hôte par glucuronoconjugaison, produisant un conjugué inactif (SN-38G). Ce métabolite est excrété dans l’intestin où le glucuronide est éliminé par les enzymes bactériennes intestinales β-glucuronidases (GUS). Cette réactivation produit une toxicité dose-limitante au niveau du tube digestif. Le groupe de Redinbo a utilisé le criblage à haut débit pour identifier des inhibiteurs sélectifs des enzymes bactériennes intestinales GUS, et il a montré que ceux-ci empêchaient la survenue des effets indésirables sévères causés par l’irinotécan dans un modèle murin [9]. Des travaux ultérieurs ont révélé que ces molécules augmentaient l’efficacité de l’irinotécan en limitant sa toxicité [10]. Ces travaux ont fourni une preuve très intéressante du concept du ciblage thérapeutique du métabolisme bactérien intestinal et sont à l’origine d’autres initiatives pour découvrir des inhibiteurs.
On estime que 70 % des microorganismes intestinaux n’ont pas encore été cultivés et il est donc difficile de caractériser leurs activités. Donia et son équipe ont utilisé la métagénomique fonctionnelle, qui consiste introduire dans un hôte hétérologue de l’ADN isolé directement dans un microbiote complexe, afin d’identifier une enzyme bactérienne intestinale métabolisant l’hydrocortisone [6]. Des enzymes métabolisant le cholestérol ont aussi été découvertes récemment dans des bactéries intestinales non cultivées en corrélant la présence de gênes microbiens dans les microbiomes aux données métabolomiques [7]. Ces deux stratégies pourraient être utiles pour étudier la métabolisation des médicaments par des microorganismes
non cultivés.
Les prochaines étapes
Le développement d’inhibiteurs de GUS comme candidats thérapeutiques est une illustration de la manière dont la compréhension des mécanismes moléculaires de la métabolisation des médicaments par le microbiote intestinal pourrait bénéficier aux patients. Un autre domaine qui pourrait être transformé par ces connaissances est la médecine de précision. En effet, en comprenant comment des traitements spécifiques sont métabolisés par les micro- organismes intestinaux, les médecins pourraient un jour utiliser les données de séquençage du microbiome ou des tests diagnostiques basés sur le microbiote pour décider si, et de quelle manière, il convient de prescrire des médicaments particuliers.
La reconnaissance croissante du rôle de la métabolisation des médicaments par le microbiote intestinal pourrait également influer sur le processus-même de découverte des médicaments. En raison de leurs associations passées avec des toxicités et des effets indésirables, de nombreux groupes fonctionnels dont on sait qu’ils sont transformés par les bactéries intestinales sont typiquement évités par les chimistes médicinaux. On pourrait imaginer découvrir de nouvelles transformations inattendues dans les premières phases du développement des médicaments, en criblant des micro-organismes intestinaux individuels ou des communautés complexes de patients à des fins de détermination du métabolisme ex vivo, de la même manière que l’on teste typiquement les candidats-médicaments afin d’analyser leur métabolisation par les enzymes de l’hôte. Les différences entre les modèles animaux et les êtres humains en termes de composition et de fonctions du microbiote intestinal devraient être prises en compte dans les études précliniques et cliniques. Enfin, il pourrait être opportun d’intégrer dans les essais cliniques le recueil d’échantillons de microbiote et leur analyse en termes de métabolisme du médicament. La mise en corrélation du métabolisme avec les différences au niveau de la toxicité ou de l’efficacité pourrait aider à interpréter les résultats de ces essais et à définir les populations de patients cibles.
Conclusion
En résumé, ces 10 dernières années ont été le témoin d’avancées considérables dans la compréhension des mécanismes moléculaires de la métabolisation des médicaments par le microbiote intestinal et de ses conséquences sur l’efficacité des médicaments. Des travaux supplémentaires pour explorer ce domaine de recherche passionnant pourraient permettre de faire avancer la médecine de précision et la découverte des médicaments.